转自:JGG 遗传学报
JGG · Research · 简讯
生物分子标志物开发是临床和转化医学研究领域的重要任务之一。利用微阵列芯片、测序等技术的组学检测已经成为发现分子标志物的主流策略。然而,受限于研究规模和成本,这类研究的初始样本量一般较小。相较于组学数据的高特征变量数,筛选的统计检验效力具有局限性,导致筛选的标志物数目远大于预期,影响下一步的研究和转化应用。LASSO算法常常作为此类研究中标志物筛选的主要方法,但其性能表现仍有很大的提升空间。2023年1月3日,Journal of Genetics and Genomics在线发表中山大学附属肿瘤防治中心生物信息平台题为“VSOLassoBag: a variable-selection oriented LASSO bagging algorithm for biomarker discovery in omic-based translational research”的研究论文。该研究推出的VSOLassoBag算法,通过整合集成学习与LASSO算法,有效提高了原有LASSO算法在生物标志物筛选任务中的性能表现。

该研究开发了VSOLassoBag算法,整合了bagging集成学习和LASSO算法,可实现从有限样本的组学数据中高效、稳定地筛选高可信度的生物标志物。基于模拟数据与真实数据的评估,该研究横向比较了VSOLassoBag与LASSO、其他集成学习类LASSO算法以及非线性机器学习算法,发现VSOLassoBag具有较佳的筛选效果,可基于基因组、转录组、表观组学等高维组学数据,筛选得到更核心的诊断和预后标志物。
为了方便用户使用,VSOLassoBag算法以R包的形式在CRAN平台、GitHub平台上进行了发布(GitHub链接:https://github.com/likelet/VSOLassoBag)。
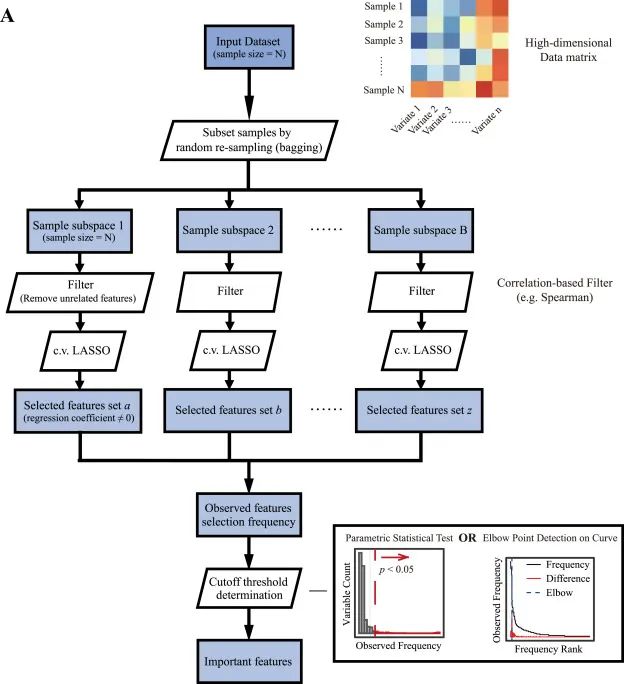
VSOLassoBag算法原理图
中山大学附属肿瘤防治中心硕士生梁嘉祺、硕士生王朝晔和中山大学附属第六医院Zhang Di博士为该论文共同第一作者,中山大学肿瘤防治中心生物信息平台任间教授团队的赵齐副研究员为该研究通讯作者。相关工作得到国家自然科学基金和广东省科技厅科研项目等资助。
Jiaqi Liang, Chaoye Wang, Di Zhang, Yubin Xie, Yanru Zeng, Tianqin Li, Zhixiang Zuo, Jian Ren, Qi Zhao. (2023). VSOLassoBag: a variable-selection oriented LASSO bagging algorithm for biomarker discovery in omic-based translational research. Journal of Genetics and Genomics.
DOI:10.1016/j.jgg.2022.12.005
(该文是JGG的快速通道论文,于2022年12月25日收稿,12月26日接收,2023年1月3日在线发表)







